化工学报 ›› 2023, Vol. 74 ›› Issue (5): 2111-2122.DOI: 10.11949/0438-1157.20221582
赵春雷1,2( ), 郭亮1,2, 高聪1,2, 宋伟3, 吴静3, 刘佳1,2, 刘立明1,2, 陈修来1,2(
), 郭亮1,2, 高聪1,2, 宋伟3, 吴静3, 刘佳1,2, 刘立明1,2, 陈修来1,2( )
)
收稿日期:2022-12-08
修回日期:2023-02-17
出版日期:2023-05-05
发布日期:2023-06-29
通讯作者:
陈修来
作者简介:赵春雷(1997—),男,硕士研究生,1972630117@qq.com
基金资助:
Chunlei ZHAO1,2( ), Liang GUO1,2, Cong GAO1,2, Wei SONG3, Jing WU3, Jia LIU1,2, Liming LIU1,2, Xiulai CHEN1,2(
), Liang GUO1,2, Cong GAO1,2, Wei SONG3, Jing WU3, Jia LIU1,2, Liming LIU1,2, Xiulai CHEN1,2( )
)
Received:2022-12-08
Revised:2023-02-17
Online:2023-05-05
Published:2023-06-29
Contact:
Xiulai CHEN
摘要:
目前生物法合成软骨素的研究策略主要侧重于构建合成路径,缺乏对前体物质供应的精细化调控,从而限制了软骨素的合成效率。为了解决上述问题,本研究借助代谢工程策略,在大肠杆菌(Escherichia coli)中重构与优化了软骨素合成路径,获得了合成软骨素的微生物细胞工厂,实现了发酵法生产软骨素。通过在E. coli BL21 STAR (DE3)中表达UDP-葡萄糖脱氢酶(KfoF)、UDP-氨基葡萄糖异构酶(KfoA)和软骨素聚合酶(KfoC),构建了完整的软骨素合成路径。通过氨基转移酶(GlmS)和磷酸葡萄糖胺变位酶(GlmM)的基因表达水平优化,提高了软骨素合成前体UDP-GalNAc的供给量;优化KfoF的基因表达水平,改善了软骨素合成前体UDP-GlcA的供给效率。在5 L发酵罐上,最优工程菌株E. coli GZ17的软骨素产量达到了2.95 g/L。上述研究策略为硫酸软骨素菌株的构建与应用奠定了基础,也为代谢工程改造生产其他糖胺聚糖提供了借鉴。
中图分类号:
赵春雷, 郭亮, 高聪, 宋伟, 吴静, 刘佳, 刘立明, 陈修来. 代谢工程改造大肠杆菌生产软骨素[J]. 化工学报, 2023, 74(5): 2111-2122.
Chunlei ZHAO, Liang GUO, Cong GAO, Wei SONG, Jing WU, Jia LIU, Liming LIU, Xiulai CHEN. Metabolic engineering of Escherichia coli for chondroitin production[J]. CIESC Journal, 2023, 74(5): 2111-2122.
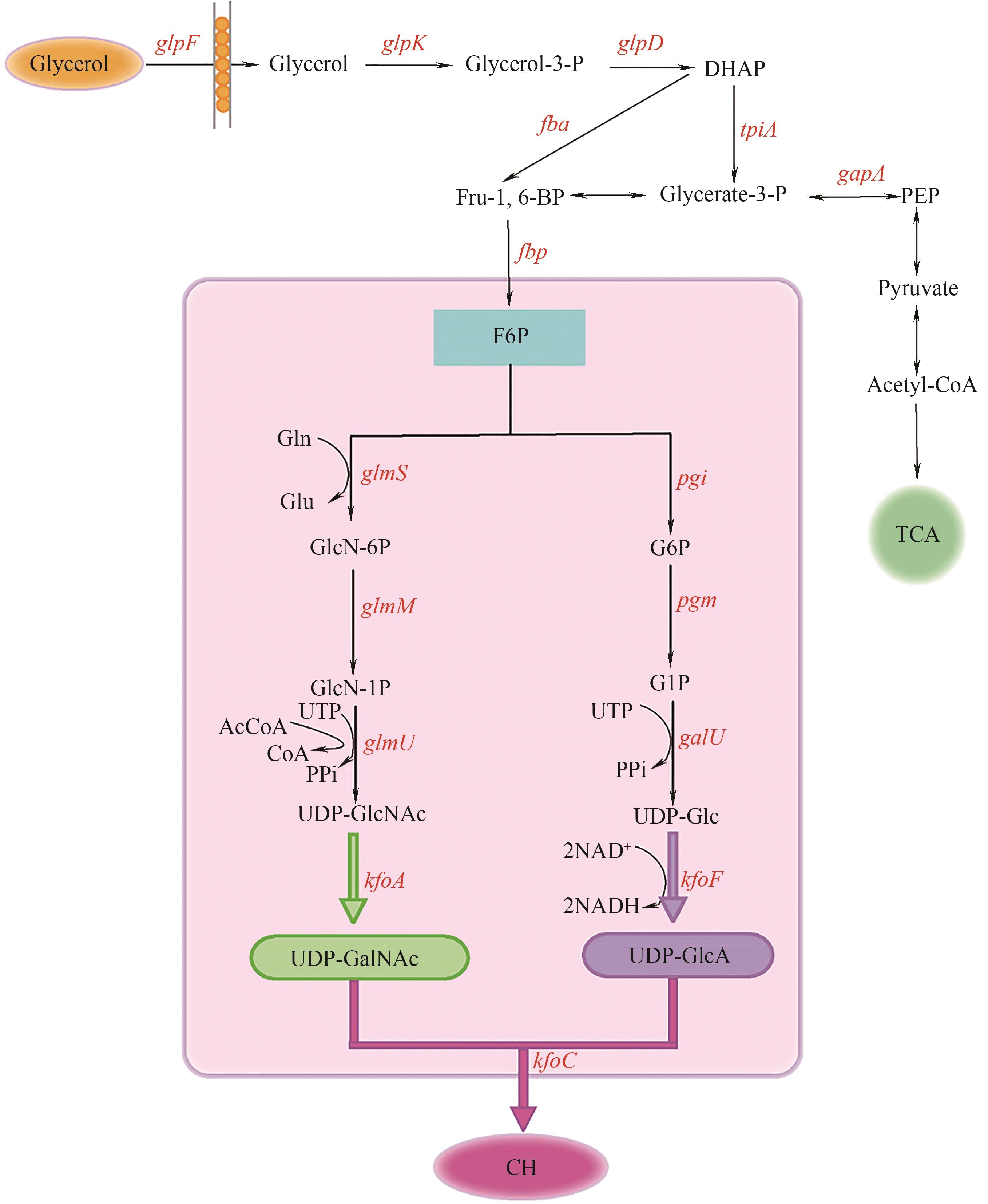
图1 大肠杆菌合成软骨素的代谢路径glmS—氨基转移酶编码基因;glmM—磷酸葡萄糖胺变位酶编码基因;glmU—UDP-N-乙酰氨基葡萄糖焦磷酸化酶编码基因;kfoA—UDP-氨基葡萄糖异构酶编码基因;pgi—葡萄糖-6-磷酸异构酶编码基因;pgm—葡萄糖磷酸变位酶编码基因;galU—葡萄糖-1-磷酸尿苷酸转移酶编码基因;kfoF—UDP-葡萄糖脱氢酶编码基因;kfoC—软骨素聚合酶编码基因;F6P—6-磷酸果糖;G6P—6-磷酸葡萄糖;G1P—1-磷酸葡萄糖;UDP-Glc—UDP-葡萄糖;UDP-GlcA—UDP-葡萄糖醛酸;GlcN-6P—6-磷酸-N-乙酰氨基葡萄糖;GlcN-1P—1-磷酸-N-乙酰氨基葡萄糖;UDP-GlcNAc—UDP-N-乙酰氨基葡萄糖;UDP-GalNAc—UDP-N-乙酰半乳糖胺;CH—软骨素
Fig.1 Metabolic pathway for chondroitin biosynthesis in Escherichia coli
| 质粒 | 描述 | 来源 |
|---|---|---|
| pEM | T5 promoter, AmpR, f1 ori | 实验室储存 |
| pCDR | Trc promoter, StrR, CloDF13 ori | 实验室储存 |
| pEM-kfoA | pEM连接基因kfoA | 本研究构建 |
| pCDR-kfoF | pCDR连接基因kfoF | 本研究构建 |
| pEM-RBSL | pEM使用低表达强度RBS | 本研究构建 |
| pEM-RBSM | pEM使用中表达强度RBS | 本研究构建 |
| pEM-RBSH | pEM使用高表达强度RBS | 本研究构建 |
| pCDR-kfoF-kfoC-kfoA | pCDR连接基因kfoA、kfoC、kfoF | 本研究构建 |
| pEM-RBSL-glmS | pEM连接低表达强度的glmS基因 | 本研究构建 |
| pEM-RBSM-glmS | pEM连接中表达强度的glmS基因 | 本研究构建 |
| pEM-RBSH-glmS | pEM连接高表达强度的glmS基因 | 本研究构建 |
| pEM-RBSL-glmM | pEM连接低表达强度的glmM基因 | 本研究构建 |
| pEM-RBSM-glmM | pEM连接中表达强度的glmM基因 | 本研究构建 |
| pEM-RBSH-glmM | pEM连接高表达强度的glmM基因 | 本研究构建 |
| pEM-RBSL-kfoF | pEM连接低表达强度的kfoF基因 | 本研究构建 |
| pEM-RBSM-kfoF | pEM连接中表达强度的kfoF基因 | 本研究构建 |
| pEM-RBSH-kfoF | pEM连接高表达强度的kfoF基因 | 本研究构建 |
表1 研究所使用的质粒
Table 1 Plasmids used in the study
| 质粒 | 描述 | 来源 |
|---|---|---|
| pEM | T5 promoter, AmpR, f1 ori | 实验室储存 |
| pCDR | Trc promoter, StrR, CloDF13 ori | 实验室储存 |
| pEM-kfoA | pEM连接基因kfoA | 本研究构建 |
| pCDR-kfoF | pCDR连接基因kfoF | 本研究构建 |
| pEM-RBSL | pEM使用低表达强度RBS | 本研究构建 |
| pEM-RBSM | pEM使用中表达强度RBS | 本研究构建 |
| pEM-RBSH | pEM使用高表达强度RBS | 本研究构建 |
| pCDR-kfoF-kfoC-kfoA | pCDR连接基因kfoA、kfoC、kfoF | 本研究构建 |
| pEM-RBSL-glmS | pEM连接低表达强度的glmS基因 | 本研究构建 |
| pEM-RBSM-glmS | pEM连接中表达强度的glmS基因 | 本研究构建 |
| pEM-RBSH-glmS | pEM连接高表达强度的glmS基因 | 本研究构建 |
| pEM-RBSL-glmM | pEM连接低表达强度的glmM基因 | 本研究构建 |
| pEM-RBSM-glmM | pEM连接中表达强度的glmM基因 | 本研究构建 |
| pEM-RBSH-glmM | pEM连接高表达强度的glmM基因 | 本研究构建 |
| pEM-RBSL-kfoF | pEM连接低表达强度的kfoF基因 | 本研究构建 |
| pEM-RBSM-kfoF | pEM连接中表达强度的kfoF基因 | 本研究构建 |
| pEM-RBSH-kfoF | pEM连接高表达强度的kfoF基因 | 本研究构建 |
| 菌株 | 描述 | 来源 |
|---|---|---|
| E. coli JM109 | Wild-type E. coli strain | 实验室储存 |
| E. coli BL21 STAR (DE3)(简称STAR) | Wild-type E. coli strain | Addgene公司购买 |
| E. coli GZ00 | STAR带有重组质粒pCDR-kfoF-kfoC-kfoA | 本研究构建 |
| E. coli GZ01 | STAR基因组整合kfoF-kfoC-kfoA | 本研究构建 |
| E. coli GZ02 | GZ00带有重组质粒pEM-RBSL-glmS | 本研究构建 |
| E. coli GZ03 | GZ00带有重组质粒pEM-RBSM-glmS | 本研究构建 |
| E. coli GZ04 | GZ00带有重组质粒pEM-RBSH-glmS | 本研究构建 |
| E. coli GZ05 | GZ01增加一个拷贝glmS | 本研究构建 |
| E. coli GZ06 | GZ01增加两个拷贝glmS | 本研究构建 |
| E. coli GZ07 | GZ01增加三个拷贝glmS | 本研究构建 |
| E. coli GZ08 | GZ00带有重组质粒pEM-RBSL-glmM | 本研究构建 |
| E. coli GZ09 | GZ00带有重组质粒pEM-RBSM-glmM | 本研究构建 |
| E. coli GZ10 | GZ00带有重组质粒pEM-RBSH-glmM | 本研究构建 |
| E. coli GZ11 | GZ06增加一个拷贝glmM | 本研究构建 |
| E. coli GZ12 | GZ06增加两个拷贝glmM | 本研究构建 |
| E. coli GZ13 | GZ06增加三个拷贝glmM | 本研究构建 |
| E. coli GZ14 | GZ00带有重组质粒pEM-RBSL-kfoF | 本研究构建 |
| E. coli GZ15 | GZ00带有重组质粒pEM-RBSM-kfoF | 本研究构建 |
| E. coli GZ16 | GZ00带有重组质粒pEM-RBSH-kfoF | 本研究构建 |
| E. coli GZ17 | GZ12增加一个拷贝kfoF | 本研究构建 |
| E. coli GZ18 | GZ12增加两个拷贝kfoF | 本研究构建 |
表2 研究所使用的菌株
Table 2 Strains used in the study
| 菌株 | 描述 | 来源 |
|---|---|---|
| E. coli JM109 | Wild-type E. coli strain | 实验室储存 |
| E. coli BL21 STAR (DE3)(简称STAR) | Wild-type E. coli strain | Addgene公司购买 |
| E. coli GZ00 | STAR带有重组质粒pCDR-kfoF-kfoC-kfoA | 本研究构建 |
| E. coli GZ01 | STAR基因组整合kfoF-kfoC-kfoA | 本研究构建 |
| E. coli GZ02 | GZ00带有重组质粒pEM-RBSL-glmS | 本研究构建 |
| E. coli GZ03 | GZ00带有重组质粒pEM-RBSM-glmS | 本研究构建 |
| E. coli GZ04 | GZ00带有重组质粒pEM-RBSH-glmS | 本研究构建 |
| E. coli GZ05 | GZ01增加一个拷贝glmS | 本研究构建 |
| E. coli GZ06 | GZ01增加两个拷贝glmS | 本研究构建 |
| E. coli GZ07 | GZ01增加三个拷贝glmS | 本研究构建 |
| E. coli GZ08 | GZ00带有重组质粒pEM-RBSL-glmM | 本研究构建 |
| E. coli GZ09 | GZ00带有重组质粒pEM-RBSM-glmM | 本研究构建 |
| E. coli GZ10 | GZ00带有重组质粒pEM-RBSH-glmM | 本研究构建 |
| E. coli GZ11 | GZ06增加一个拷贝glmM | 本研究构建 |
| E. coli GZ12 | GZ06增加两个拷贝glmM | 本研究构建 |
| E. coli GZ13 | GZ06增加三个拷贝glmM | 本研究构建 |
| E. coli GZ14 | GZ00带有重组质粒pEM-RBSL-kfoF | 本研究构建 |
| E. coli GZ15 | GZ00带有重组质粒pEM-RBSM-kfoF | 本研究构建 |
| E. coli GZ16 | GZ00带有重组质粒pEM-RBSH-kfoF | 本研究构建 |
| E. coli GZ17 | GZ12增加一个拷贝kfoF | 本研究构建 |
| E. coli GZ18 | GZ12增加两个拷贝kfoF | 本研究构建 |
| 菌株 | OD600 | CH/ (mg/L) | CH对细胞得率/ (mg/g) | CH对甘油得率/ (mg/g) |
|---|---|---|---|---|
| GZ01 | 11.0 | 7.1 | 1.6 | 0.5 |
| GZ05 | 11.2 | 29.9 | 6.5 | 2.0 |
| GZ06 | 11.8 | 46.2 | 9.5 | 3.1 |
| GZ07 | 11.2 | 45.1 | 9.8 | 3.0 |
| GZ11 | 12.2 | 54.4 | 10.8 | 3.6 |
| GZ12 | 12.8 | 80.1 | 15.2 | 5.3 |
| GZ13 | 12.2 | 79.9 | 15.9 | 5.3 |
| GZ17 | 12.1 | 108.8 | 21.8 | 7.3 |
| GZ18 | 11.6 | 70.7 | 14.8 | 4.7 |
表3 软骨素合成菌株的发酵参数比较
Table 3 Comparison of fermentation parameters of chondroitin synthesis strains
| 菌株 | OD600 | CH/ (mg/L) | CH对细胞得率/ (mg/g) | CH对甘油得率/ (mg/g) |
|---|---|---|---|---|
| GZ01 | 11.0 | 7.1 | 1.6 | 0.5 |
| GZ05 | 11.2 | 29.9 | 6.5 | 2.0 |
| GZ06 | 11.8 | 46.2 | 9.5 | 3.1 |
| GZ07 | 11.2 | 45.1 | 9.8 | 3.0 |
| GZ11 | 12.2 | 54.4 | 10.8 | 3.6 |
| GZ12 | 12.8 | 80.1 | 15.2 | 5.3 |
| GZ13 | 12.2 | 79.9 | 15.9 | 5.3 |
| GZ17 | 12.1 | 108.8 | 21.8 | 7.3 |
| GZ18 | 11.6 | 70.7 | 14.8 | 4.7 |
| 发酵参数 | E. coli GZ01 | E. coli GZ17 |
|---|---|---|
| 起始OD600 | 0.47 | 0.45 |
| 最终OD600 | 57.00 | 76.00 |
| 软骨素产量/(g/L) | 0.31 | 2.95 |
| 软骨素对细胞得率/(mg/g) | 13.20 | 94.20 |
| 软骨素对甘油得率/(mg/g) | 2.25 | 13.41 |
| 生产强度/(mg/(L·h)) | 6.20 | 59.00 |
表4 E. coli GZ01和E. coli GZ17发酵结果分析
Table 4 Analysis of fermentation results of E. coli GZ01 and E. coli GZ17
| 发酵参数 | E. coli GZ01 | E. coli GZ17 |
|---|---|---|
| 起始OD600 | 0.47 | 0.45 |
| 最终OD600 | 57.00 | 76.00 |
| 软骨素产量/(g/L) | 0.31 | 2.95 |
| 软骨素对细胞得率/(mg/g) | 13.20 | 94.20 |
| 软骨素对甘油得率/(mg/g) | 2.25 | 13.41 |
| 生产强度/(mg/(L·h)) | 6.20 | 59.00 |
| 1 | Laezza A, Iadonisi A, de Castro C, et al. Chemical fucosylation of a polysaccharide: a semisynthetic access to fucosylated chondroitin sulfate[J]. Biomacromolecules, 2015, 16(7): 2237-2245. |
| 2 | Zhang X, Liu H Y, Yao W, et al. Semisynthesis of chondroitin sulfate oligosaccharides based on the enzymatic degradation of chondroitin[J]. The Journal of Organic Chemistry, 2019, 84(11): 7418-7425. |
| 3 | Legendre F, Bauge C, Roche R, et al. Chondroitin sulfate modulation of matrix and inflammatory gene expression in IL-1β-stimulated chondrocytes—study in hypoxic alginate bead cultures[J]. Osteoarthritis and Cartilage, 2008, 16(1): 105-114. |
| 4 | Maruyama T, Toida T, Imanari T, et al. Conformational changes and anticoagulant activity of chondroitin sulfate following its O-sulfonation[J]. Carbohydrate Research, 1998, 306(1/2): 35-43. |
| 5 | Martins J R M, Gadelha M E C, Fonseca S M, et al. Patients with head and neck tumors excrete a chondroitin sulfate with a low degree of sulfation: a new tool for diagnosis and follow-up of cancer therapy[J]. Otolaryngology-Head and Neck Surgery, 2000, 122(1): 115-118. |
| 6 | Volpi N. Chondroitin sulfate safety and quality[J]. Molecules, 2019, 24(8): 1447. |
| 7 | Couto M R, Rodrigues J L, Rodrigues L R. Heterologous production of chondroitin[J]. Biotechnology Reports, 2022, 33: e00710. |
| 8 | Shi Y G, Meng Y C, Li J R, et al. Chondroitin sulfate: extraction, purification, microbial and chemical synthesis[J]. Journal of Chemical Technology & Biotechnology, 2014, 89(10): 1445-1465. |
| 9 | Abdallah M M, Fernandez N, Matias A A, et al. Hyaluronic acid and chondroitin sulfate from marine and terrestrial sources: extraction and purification methods[J]. Carbohydrate Polymers, 2020, 243: 116441. |
| 10 | Xu S Q, Qiu M L, Zhang Q, et al. Chain structure and immunomodulatory activity of a fructosylated chondroitin from an engineered Escherichia coli K4[J]. International Journal of Biological Macromolecules, 2019, 133: 702-711. |
| 11 | Badri A, Williams A, Awofiranye A, et al. Complete biosynthesis of a sulfated chondroitin in Escherichia coli [J]. Nature Communications, 2021, 12: 1389. |
| 12 | Kobayashi S, Morii H, Itoh R, et al. Enzymatic polymerization to artificial hyaluronan: a novel method to synthesize a glycosaminoglycan using a transition state analogue monomer[J]. Journal of the American Chemical Society, 2001, 123(47): 11825-11826. |
| 13 | Li J E, Sparkenbaugh E M, Su G W, et al. Enzymatic synthesis of chondroitin sulfate E to attenuate bacteria lipopolysaccharide-induced organ damage[J]. ACS Central Science, 2020, 6(7): 1199-1207. |
| 14 | Li J E, Su G W, Liu J. Enzymatic synthesis of homogeneous chondroitin sulfate oligosaccharides[J]. Angewandte Chemie, 2017, 56(39): 11784-11787. |
| 15 | Sugiura N, Shimokata S, Minamisawa T, et al. Sequential synthesis of chondroitin oligosaccharides by immobilized chondroitin polymerase mutants[J]. Glycoconjugate Journal, 2008, 25(6): 521-530. |
| 16 | Gottschalk J, Zaun H, Eisele A, et al. Key factors for A one-pot enzyme cascade synthesis of high molecular weight hyaluronic acid[J]. International Journal of Molecular Sciences, 2019, 20(22): 5664. |
| 17 | Zhang Q, Yao R, Chen X L, et al. Enhancing fructosylated chondroitin production in Escherichia coli K4 by balancing the UDP-precursors[J]. Metabolic Engineering, 2018, 47: 314-322. |
| 18 | Bedini E, De Castro C, De Rosa M, et al. A microbiological-chemical strategy to produce chondroitin sulfate A, C[J]. Angewandte Chemie, 2011, 50(27): 6160-6163. |
| 19 | Cimini D, Restaino O F, Catapano A, et al. Production of capsular polysaccharide from Escherichia coli K4 for biotechnological applications[J]. Applied Microbiology and Biotechnology, 2010, 85(6): 1779-1787. |
| 20 | Restaino O F, Cimini D, De Rosa M, et al. High-performance CE of Escherichia coli K4 cell surface polysaccharides[J]. Electrophoresis, 2009, 30(22): 3877-3883. |
| 21 | He W Q, Fu L, Li G Y, et al. Production of chondroitin in metabolically engineered E. coli [J]. Metabolic Engineering, 2015, 27: 92-100. |
| 22 | Cheng F Y, Luozhong S J, Yu H M, et al. Biosynthesis of chondroitin in engineered Corynebacterium glutamicum [J]. Journal of Microbiology and Biotechnology, 2019, 29(3): 392-400. |
| 23 | Jin P, Zhang L P, Yuan P H, et al. Efficient biosynthesis of polysaccharides chondroitin and heparosan by metabolically engineered Bacillus subtilis [J]. Carbohydrate Polymers, 2016, 140: 424-432. |
| 24 | Zhou Z X, Li Q, Huang H, et al. A microbial-enzymatic strategy for producing chondroitin sulfate glycosaminoglycans[J]. Biotechnology and Bioengineering, 2018, 115(6): 1561-1570. |
| 25 | Karim A S, Curran K A, Alper H S. Characterization of plasmid burden and copy number in Saccharomyces cerevisiae for optimization of metabolic engineering applications[J]. FEMS Yeast Research, 2013, 13(1): 107-116. |
| 26 | Kang C W, Lim H G, Yang J, et al. Synthetic auxotrophs for stable and tunable maintenance of plasmid copy number[J]. Metabolic Engineering, 2018, 48: 121-128. |
| 27 | Zhao M, Huang D, Zhang X, et al. Metabolic engineering of Escherichia coli for producing adipic acid through the reverse adipate-degradation pathway[J]. Metabolic Engineering, 2018, 47: 254-262. |
| 28 | Zhang C, Liu L, Teng L, et al. Metabolic engineering of Escherichia coli BL21 for biosynthesis of heparosan, a bioengineered heparin precursor[J]. Metabolic Engineering, 2012, 14(5): 521-527. |
| 29 | Jin X R, Zhang W J, Wang Y, et al. Biosynthesis of non-animal chondroitin sulfate from methanol using genetically engineered Pichia pastoris [J]. Green Chemistry, 2021, 23(12): 4365-4374. |
| 30 | Cimini D, Iacono I D, Carlino E, et al. Engineering S. equi subsp. zooepidemicus towards concurrent production of hyaluronic acid and chondroitin biopolymers of biomedical interest[J]. AMB Express, 2017, 7(1): 61. |
| 31 | Rouches M V, Xu Y S, Cortes L B G, et al. A plasmid system with tunable copy number[J]. Nature Communications, 2022, 13: 3908. |
| 32 | Lemuth K, Steuer K, Albermann C. Engineering of a plasmid-free Escherichia coli strain for improved in vivo biosynthesis of astaxanthin[J]. Microbial Cell Factories, 2011, 10: 29. |
| [1] | 刘雪, 张莉娟, 赵广荣. 大肠杆菌偏利共培养系统合成大豆苷元[J]. 化工学报, 2022, 73(9): 4015-4024. |
| [2] | 郑煜堃, 孙青, 陈振, 于慧敏. 微生物细胞工厂生产化学品的研究进展——以几种典型小分子和大分子化学品为例[J]. 化工学报, 2021, 72(12): 6109-6121. |
| [3] | 王凯峰, 王金鹏, 韦萍, 纪晓俊. 代谢工程改造解脂耶氏酵母生产脂肪酸及其衍生物[J]. 化工学报, 2021, 72(1): 351-365. |
| [4] | 张耀, 邱晓曼, 陈程鹏, 于卓然, 洪厚胜. 生物法制造丁二酸研究进展[J]. 化工学报, 2020, 71(5): 1964-1975. |
| [5] | 徐彦芹, 杨锡智, 罗若诗, 黄玉红, 霍锋, 王丹. 合成生物学在生物基塑料制造中的应用[J]. 化工学报, 2020, 71(10): 4520-4531. |
| [6] | 陈天华, 张若思, 姜国珍, 姚明东, 刘宏, 王颖, 肖文海, 元英进. 产蒎烯人工酵母细胞的构建[J]. 化工学报, 2019, 70(1): 179-188. |
| [7] | 武耀康, 刘延峰, 李江华, 堵国成, 刘龙, 陈坚. 动态调控元件及其在微生物代谢工程中的应用[J]. 化工学报, 2018, 69(1): 272-281. |
| [8] | 王乐, 倪子富, 惠明, 王金水. 代谢控制发酵产琥珀酸研究进展[J]. 化工学报, 2015, 66(4): 1243-1251. |
| [9] | 皇甫洁, 李茜, 李珺, 李春. 模拟微重力下影响重组Escherichia coli外源蛋白质表达途径分析[J]. 化工学报, 2015, 66(12): 4960-4965. |
| [10] | 赵雨佳, 张根林, 周晓宏, 李春. 核糖核酸开关用于微生物细胞工厂的智能与精细调控[J]. 化工学报, 2015, 66(10): 3811-2819. |
| [11] | 孙翔英, 刘月芹, 孙欢, 贾海洋, 戴大章, 李春. 大肠杆菌耐热元器件的构建及其应用[J]. 化工学报, 2014, 65(8): 3128-3135. |
| [12] | 苏楠, 吴敬君, 李晔, 张翀, 李梅, 邢新会. MBP融合肝素酶Ⅲ大肠杆菌高效表达体系构建[J]. 化工学报, 2014, 65(7): 2829-2842. |
| [13] | 马强强,赵广荣. 左旋多巴合成研究进展[J]. 化工进展, 2013, 32(06): 1367-1371. |
| [14] | 茅羽佳,欧先金,杜伟,刘德华. 大肠杆菌高水平表达脂肪酶BTL2及其催化油脂制备生物柴油[J]. 化工学报, 2012, 63(7): 2100-2105. |
| [15] | 于化江1,熊 亮2,熊中琼2,张国庆2. 复合电沉积制备TiO2/泡沫镍光催化材料及其催化活性 [J]. CIESC Journal, 2011, 30(9): 1972-. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||
 京公网安备 11010102001995号
京公网安备 11010102001995号